La hibridación in situ permite detectar la presencia de una secuencia de nucleótidos en una célula. Esto se consigue mediante otra secuencia de nucleótidos denominada sonda que es complementaria a la secuencia que queremos identificar. La complementariedad entre secuencias de bases nucleotídicas es la base de la especificidad de esta técnica. Así, podemos descubrir qué células expresan un gen determinado y cuándo lo expresan, incluso la intensidad con que lo hacen. La expresión génica es un testimonio directo de la funcionalidad celular. Existe otra aplicación de la hibridación in situ que consiste en la localización física de un gen sobre un cromosoma. No se puede considerar a la hibridación in situ como una técnica de uso general. Sin embargo, aporta una información sobre la fisiología de las células y los tejidos que no es posible obtener con otras técnicas. Se puede emplear en células, tejidos, o animales enteros, normalmente embriones. La hibridación in situ se usa ampliamente en investigación en desarrollo embrionario, diferenciación de células madre, manipulaciones genéticas o cambios en la fisiología celular frente a señales externas, entre otras.
Preparación del tejido
Cuando se hace sobre tejidos, órganos o embriones, hay que tratar el tejido previamente antes de añadir la sonda. Como siempre, los tejidos han de estar fijados, siendo el formaldehído el fijador más frecuente. Además, cuando hay que trabajar con secciones, es preferible congelar y obtener secciones por congelación, en vez de incluir en parafina, puesto que el ARN se preserva mejor. Tras la fijación, crioprotección y congelación, el material se puede mantener congelado durante largo tiempo.
En el caso de detección de ARN es importante tener en cuenta que es una molécula que se degrada con facilidad, por lo que tenemos que tomar medidas para no perderla. El ARN se degrada por las ARNasas, y una gran cantidad de este enzima se encuentra en nuestras manos, por lo que tendremos que usar guantes y todo el material que se use ha de estar libre de RNAsas (con un autoclavado se pueden inactivar). A los tejidos se les trata antes para prepararlos para favorecer la penetración de la sonda, por ejemplo, con proteasas. También la acetilación (ácido acético en tampón trietanolamina) disminuye las uniones del sonda de manera inespecífica al tejido.
Sonda
La hibridación in situ se basa en la complementariedad de bases nucleotídicas (A-T o A-U y G-C). Del mismo modo que en la inmunocitoquímica se usan los anticuerpos, en la hibridación in situ se usa una secuencia de nucleótidos, denominada sonda, que es complementaria a la secuencia del ARN que queremos detectar, y que está conjugada con una molécula que luego pondremos de manifiesto y que nos sirve de marcador. El tamaño apropiado de una sonda es de entre 50 a 300 bases. Las sondas se pueden marcar de manera radiactiva (isotópica) o no radiactiva (no isotópica). Esta última es la más frecuente. Las no isotópicas se pueden marcar con biotina, sustancias fluorescentes, digoxigenina, bromodeoxiuridina, y otras sustancias.
Quizá una de las razones por las que la hibridación in situ no es común todavía en los laboratorios de histología es porque sintetizar una sonda es complicado y generalmente no se puede usar en una especie animal o vegetal distinta a aquella para la que se ha producido dicha sonda. Ello es porque un mismo gen (y su ARN mensajero) en dos especies distintas tienen secuencias que no son idénticas y por tanto hay que fabricar una sonda para cada especie. Sin embargo, una vez obtenida la sonda el proceso de hibridación es sencillo.
Para obtener una sonda lo primero que tenemos que hacer es conocer la secuencia de bases del ARNm con el que queremos hibridarla. Si esa secuencia está publicada en las bases de datos en Internet todo el proceso se simplifica enormemente puesto que hoy en día se puede solicitar la síntesis de forma química de secuencias largas de ADN (hasta 1000 nucleótidos es común) a empresas especializadas. A partir de ese ADN podemos obtener nuestras sondas en el laboratorio (ver más abajo). Pero incluso, se pueden comprar las sondas ya marcadas, con lo que nos ahorramos una gran cantidad de tiempo en el laboratorio. Actualmente hay bases de datos enormes con multitud de genes clonados con sus secuencias publicadas a partir de las cuales se pueden obtener las sondas. Además, los principales especies modelo de estudio, tanto en animales como en vegetales, tienen sus genomas completos secuenciados por lo que podremos buscar en dichos genomas la secuencia génica en la que estemos interesados.
Sin embargo, en muchos casos no conocemos la secuencia de bases del gen deseado por lo que hay que averiguarlo primero para obtener posteriormente la sonda. Para obtener una sonda hay primero que clonar la secuencia de bases del ARN mensajero que queremos detectar. Clonar implica realizar una serie de pasos. 1) Una vez purificado el ARN de nuestro tejido, los ARN mensajeros se retrotranscriben (transcripción inversa), es decir, se hacen copias complementarias de ADN de todos los fragmentos de ARN mensajero. Se obtienen hebras de ADN denominadas cDNA (ADN clonado). 2) Mediante la técnica de PCR ("polymerase chain reaction") se consiguen multitud de copias de manera específica de la secuencia que queremos estudiar. 3) Dichas copias se integran en un plásmido y posteriormente se introduce en bacterias. 4) Se cultivan las bacterias para que proliferen y así también conseguir gran número de copias de los plásmidos, que se duplican en cada división bacteriana. 5) Los plásmidos se extraen y purifican. 6) Mediante un proceso de transcripción similar al que ocurre en el núcleo de las células, a partir de estos plásmidos se consiguen numerosas réplicas de ARN complementarias a nuestra secuencia inserta, que será también complementaria a la secuencia del ARN mensajero que queremos detectar. El resultado de la transcripción realizada repetidas veces es un gran número de cadenas de ARN denominadas sondas. Durante la síntesis de la sonda es cuando se añaden los nucleótidos conjugados con las moléculas marcadoras que nos servirán para detectarla una vez la hibridación se haya producido. Normalmente son moléculas pequeñas como la digoxigenina y la biotina, aunque también se pueden utilizar moléculas fluorescentes, que se unen a los nucleótidos que formarán parte de la sonda.
Proceso de hibridación
Una vez sintetizada la sonda marcada, se pone en contacto con el tejido e hibridará con aquellos ARN mensajeros que sean complementarios. El lugar de hibridación se pondrá de manifiesto cuando detectemos nuestro marcador unido a la sonda (por ejemplo, digoxigenina) con un anticuerpo conjugado con un enzima (por ejemplo, la fosfatasa alcalina) (Figuras 1, 2 y 3 ).
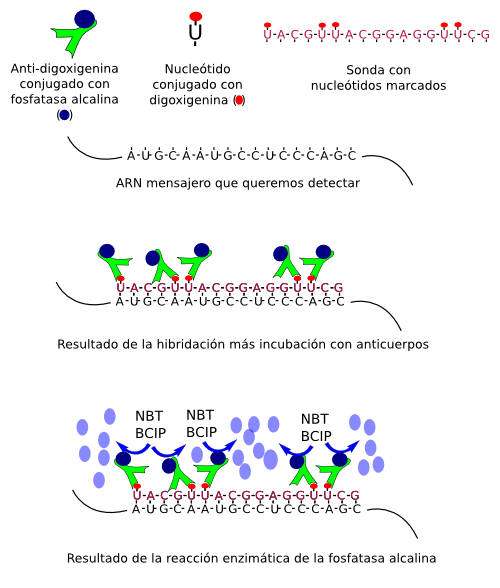
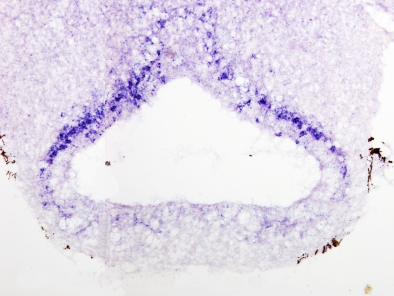
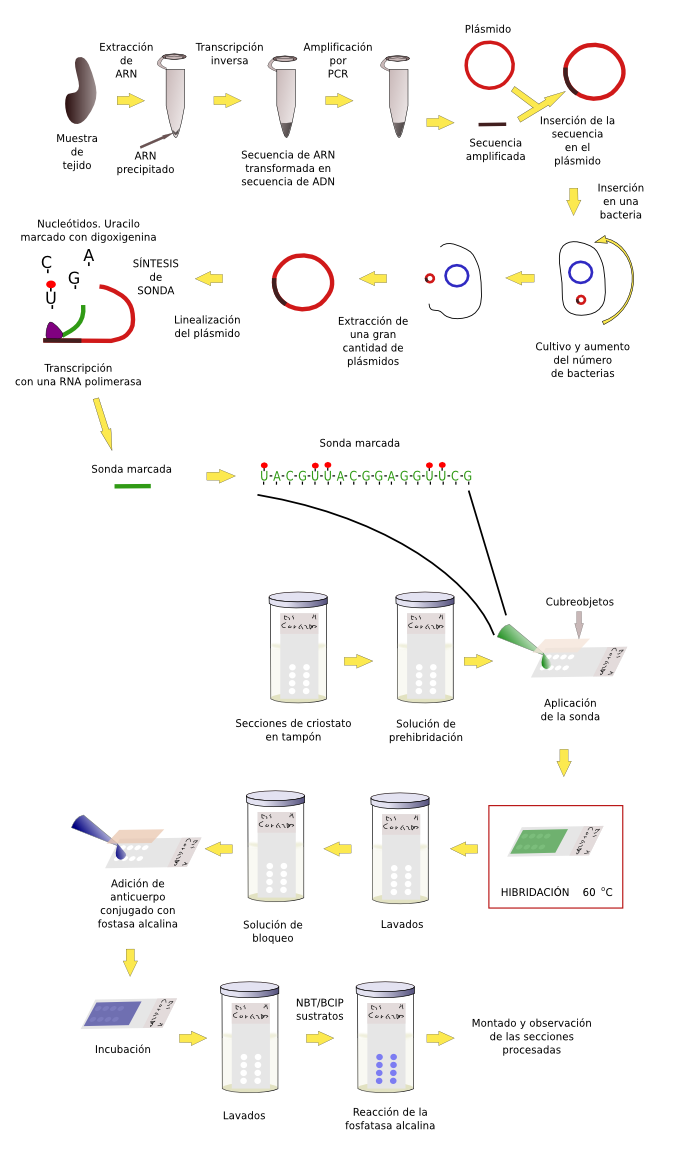
La hibridación in situ se puede combinar con otras técnicas como la inmunocitoquímica o con tinciones generales.
Una variedad de la hibridación in situ muy usada consiste en conjugar la sonda con sustancias fluorescentes (FISH, “fluorescent in situ hybridization”). Es muy utilizada para localizar genes en los cromosomas.
 Inmunohistoquímica
Inmunohistoquímica